[Molecular Plant] 华南农大夏瑞团队发表TBtools-II,为个性化生物数据分析提供解决方案
时间:2023-09-26 阅读量:1267
植物生物大数据分析是当前植物学研究的主要任务之一。为辅助更多植物科学家处理生物大数据,TBtools-I 应运而生,并于2020年08月发表于 Molecular Plant 期刊(Chen et al., 2020)。过去三年间,植物生物信息领域有长足发展,如涌现了较多物种的T2T基因组与单倍型基因组(Naish et al., 2021, Sun et al., 2022, Shang et al., 2023, Shi et al., 2023),同时伴随本地存储(固态硬盘,SSD)和计算资源(CPU甚至GPU)价格的进一步下降,更多人可以在本地电脑开展生物大数据分析,也因此带来了更多的个性化数据分析需求。TBtools-I 着力解决植物生物数据分析的共性需求,如序列操作,序列比对,基因集功能分析以及生物数据可视化等。随着软件用户群体持续增长,需求多样性丰富,TBtools-I 已有功能和模式无法良好应对(陈程杰 和 夏瑞, 2022)。
一方面,不同的用户对数据分析有着不同的需求,往往希望使用特定的工具或工作流程进行数据分析,以获得最佳结果。因此,越来越多的功能被建议纳入用户熟悉的 TBtools 软件中,以满足个性化数据分析不断增长的需求,例如来自不同测序策略(如ChIP-seq,DAP-seq,BSA-seq等)生成的不同NGS数据;另一方面,更多新功能的添加(自发布以来已新增100多个新功能),使工具包变得过于复杂和繁琐,稀释了 TBtools 最初关注的用户共性需求的功能,反过来使用户很难快速找到所需的功能。此外,作为一款桌面软件,软件体积变大会增加分发、安装和使用方面上的复杂性,进而影响整体鲁棒性。
为应对这一困境,2023年9月22日,华南农业大学园艺学院(亚热带农业生物资源保护与利用国家重点实验室)夏瑞团队在 Molecular Plant 在线发表了题为 TBtools-II: A One for All, All for One Bioinformatics Platform for Biological Big-data Mining 的研究论文,系统介绍和描述 TBtools 的新版本,即 TBtools-II。TBtools-II 在 TBtools-I 的基础上优化并新增了100+个功能,开发了插件(Plugin)模式并架设了插件仓库(Plugin Store)。基于此,解决了软件功能全面性与灵活性的主要冲突。
另外,受限于 TBtools 项目组的课题经验,绝大多数 TBtools 功能(包括项目组开发的功能)局限在常规分子生物学和基因组学领域,未能良好拓展到诸如群体遗传学,表观遗传学甚至单细胞组学等领域。最好的软件开发者往往是用户。借由插件模式和插件仓库的推出,TBtools-II 同步提供了简便的插件开发接口,使得用户可以直接依据自己的需求,灵活地开发出实用插件,继而在课题组内甚至在TBtools用户社区分享,让所有TBtools用户受益。从而使得TBtools 由“软件”模式逐步走向“平台”模式。

https://doi.org/10.1016/j.molp.2023.09.010
“开发者即用户,用户即开发者”,TBtools-II 提出“我为人人,人人为我”的开发新理念,插件商店(Plugin Store)也应运而生。为辅助用户参与开发,项目团队开发并释放了一款辅助开发 TBtools 插件的插件 - “CLI Program Wrapper Creator”。在该插件中,用户可以通过简单的表单填写,将本地二进制程序、脚本以及相关依赖转变成一个可以用于分发的 TBtools 插件。同时插件可上传到插件商店,分享给所有 TBtools 用户使用。
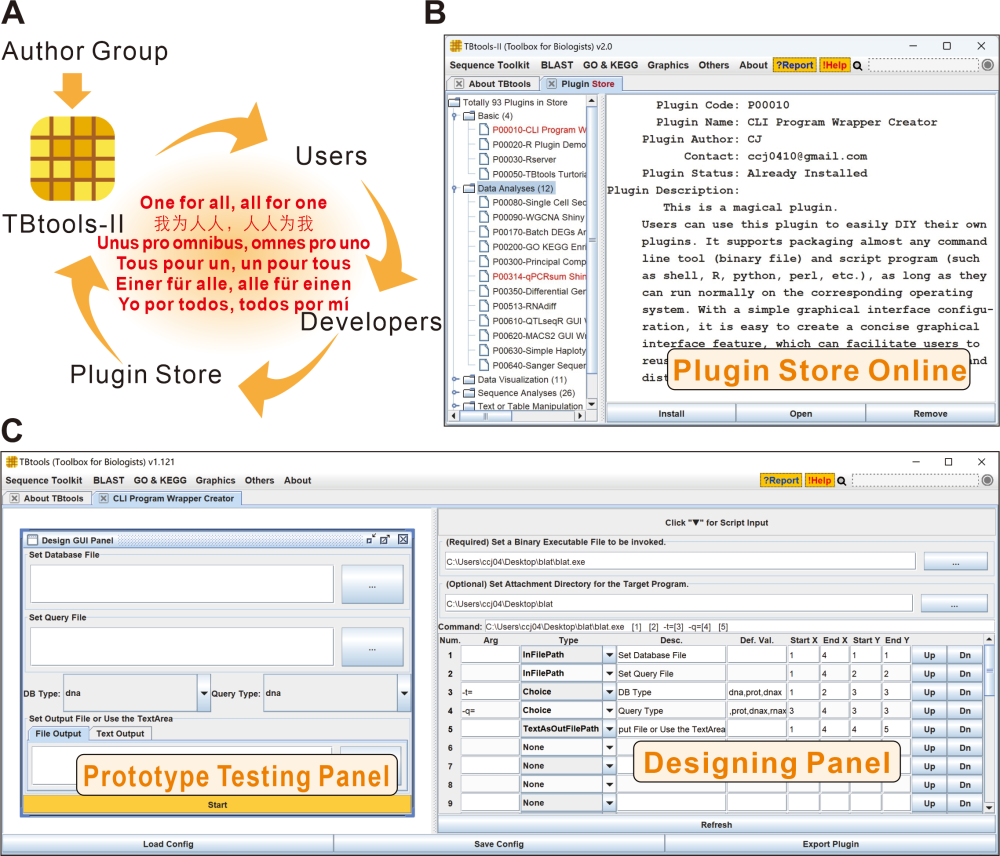
图 1. TBtools-II 以及 插件模式
当前,TBtools 插件商店中已有 94 个插件,超过半数为 TBtools 用户开发,功能覆盖了生物文本处理和数据可视化等。论文中介绍了由两位用户(受邀作为 TBtools-II 论文共同作者)开发的用户常用插件:用于植物单细胞数据分析的 Single Cell Sequencing Seurat Shiny 以及用于基因共表达网络分析的 WGCNA Shiny。
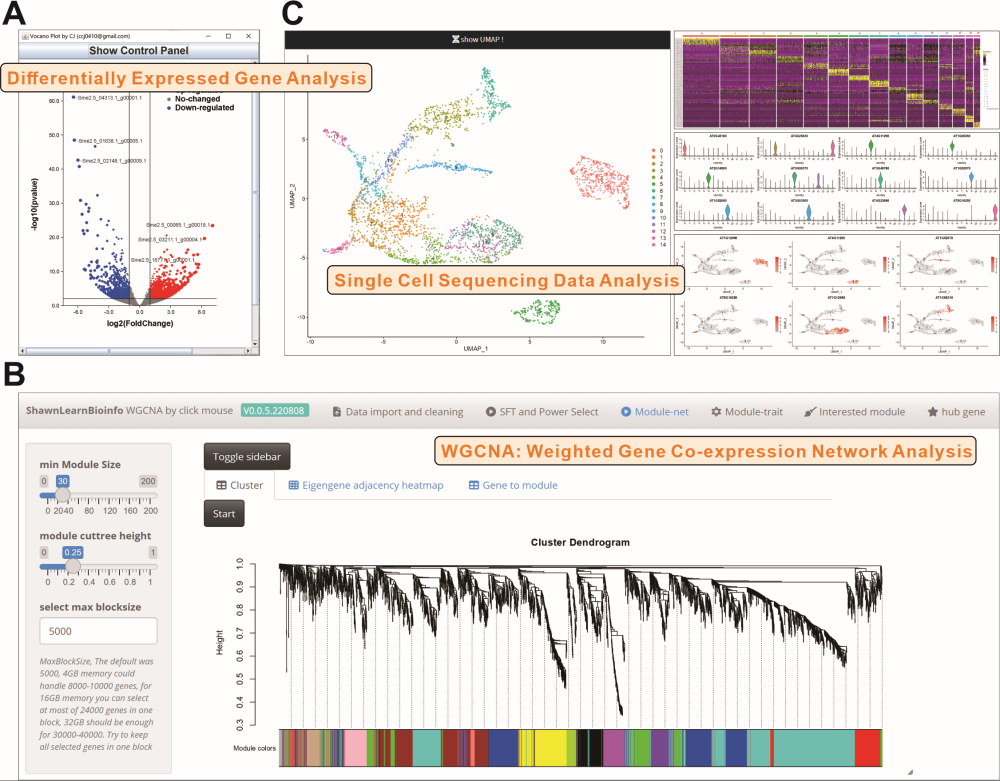
图 2. 用户常用插件功能示例(差异表达分析、单细胞数据分析以及共表达网络分析)
顺应用户生物育种工作需求,TBtools-II 同步引入了系列常见插件和功能,可以便捷完成基于比较基因组的结构变异检测、引物自动开发和电子PCR和核酸电泳模拟,支持了本地电脑重测序数据快速回帖和SNP/Indel检测,结果可同步用于 QTLs 鉴定(BSAseq)。为辅助用户更好了解相关功能,文稿给出了一个基于公开发表的番茄 BSAseq 数据(Soyk et al., 2019)的分析流程示例,详细步骤参考正文。
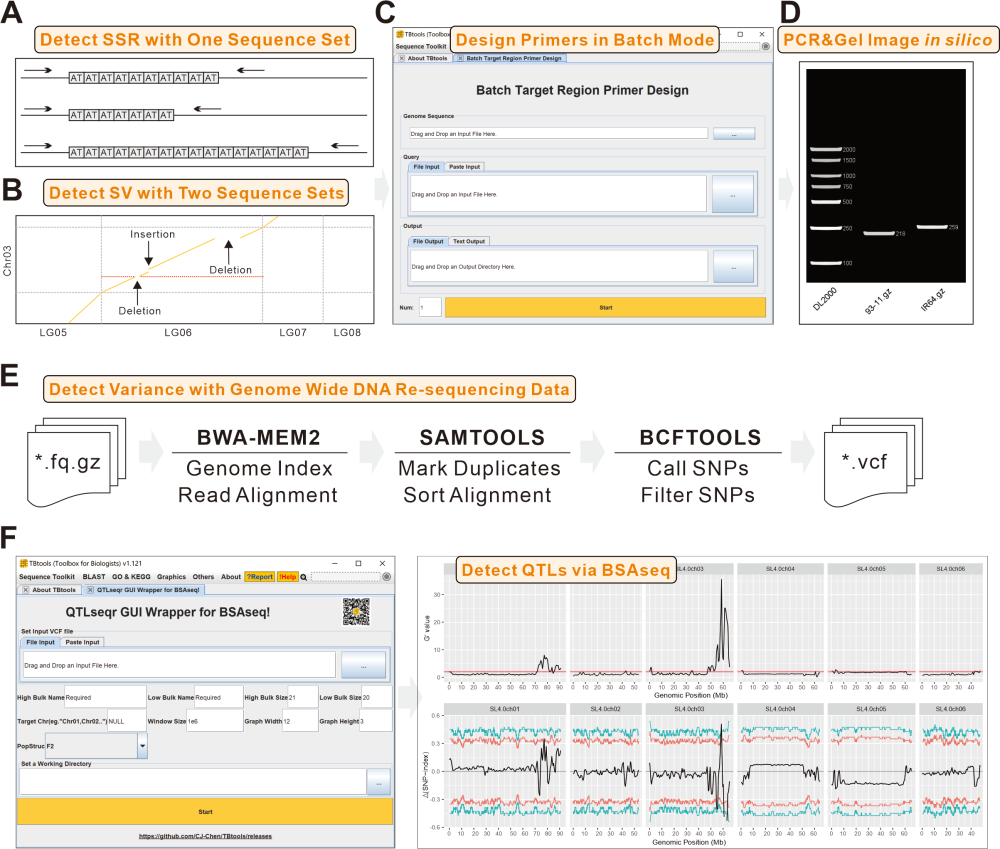
图 3. 后基因组时代的生物育种数据分析插件(分子标记快速开发与QTLseq数据分析)
华南农业大学园艺学院夏瑞教授和陈程杰讲师为论文的通讯作者,陈程杰讲师为论文第一作者;TBtools项目组(吴亚、曾灶海、徐婧、刘元龙副教授、何业华教授),暨南大学李嘉威博士,河南大学王骁博士,中国热带科学院生物所冯筠庭博士以及湖南农业大学陈浩博士参与了软件优化建议、开发测试等工作。论文工作得到广东省重点研发、国家自然科学基金、国家重点研发等项目支持,同步致谢了华南农业大学夏瑞课题组、何业华课题组、徐春香课题组、贵阳沃奇生物、邵扬(华中农业大学)、赵齐(中山大学肿瘤防治中心)和曾健明(澳门大学)等在 TBtools 插件生态构件上的建议和帮助,以及数以万计的TBtools软件用户,尤其是>40名TBtools社群管理人员的贡献。
夏瑞课题组长期关注小RNA和园艺植物性别演化规律探索,以植物基因组和生物信息学为主要研究手段,并研发了系列广为认可生物软件:植物生物信息学数据下游分析的 TBtools(Chen et al., 2020);正选择位点分析 EasyCodeML(Gao et al., 2019);植物小RNA注释数据库 sRNAanno(Chen et al., 2021);无患子科植物基因组数据库Sapbase(Li et al., 未发表);植物小RNA组学数据分析一站式软件 sRNAminer 和基因组浏览器 IGV-sRNA(Li et al., 未发表)以及基因结构人工矫正软件 IGV-GSAman(Chen et al., 未发表)以及其他。
参考文献
陈程杰,夏瑞. TBtools——大数据时代下的国产生物软件. 科学观察, 2022,17(06):33-35.
Chen, C., Chen, H., Zhang, Y., Thomas, H. R., Frank, M. H., He, Y., and Xia, R. (2020). TBtools: An Integrative Toolkit Developed for Interactive Analyses of Big Biological Data. Mol. Plant 13:1194–1202.
Chen C, Li J, Feng J, Liu B, Feng L, Yu X, Li G, Zhai J, Meyers BC, Xia R. sRNAanno-a database repository of uniformly annotated small RNAs in plants. Hortic Res. 2021 Mar 1;8(1):45
Chen Chengjie , Wu Ya , Li Jiawei, Wang Xiao , Zeng Zaohai , Xu Jing , Liu Yuanlong , Feng Junting, Chen Hao ,He Yehua , Xia Rui ,TBtools-II: A One for All, All for One Bioinformatics Platform for Biological Big-data Mining, Molecular Plant, 2023.
Gao F, Chen C, Arab DA, Du Z, He Y, Ho SYW. EasyCodeML: A visual tool for analysis of selection using CodeML. Ecol Evol. 2019 Mar 1;9(7):3891-3898.
Naish, M., Alonge, M., Wlodzimierz, P., Tock, A. J., Abramson, B. W., Schmücker, A., Mandáková, T., Jamge, B., Lambing, C., Kuo, P., et al. (2021). The genetic and epigenetic landscape of the Arabidopsis centromeres. Science 374.
Shang, L., He, W., Wang, T., Yang, Y., Xu, Q., Zhao, X., Yang, L., Zhang, H., Li, X., Lv, Y., et al. (2023). A complete assembly of the rice Nipponbare reference genome. Mol Plant Advance Access published August 2023, doi:10.1016/j.molp.2023.08.003.
Shi, X., Cao, S., Wang, X., Huang, S., Wang, Y., Liu, Z., Liu, W., Leng, X., Peng, Y., Wang, N., et al. (2023). The complete reference genome for grapevine (Vitis vinifera L.) genetics and breeding. Hortic Res 10.
Soyk, S., Lemmon, Z. H., Sedlazeck, F. J., Jiménez-Gómez, J. M., Alonge, M., Hutton, S. F., Van Eck, J., Schatz, M. C., and Lippman, Z. B. (2019). Duplication of a domestication locus neutralized a cryptic variant that caused a breeding barrier in tomato. Nat. Plants 5:471–479.
Sun, H., Jiao, W.-B., Krause, K., Campoy, J. A., Goel, M., Folz-Donahue, K., Kukat, C., Huettel, B., and Schneeberger, K. (2022). Chromosome-scale and haplotype-resolved genome assembly of a tetraploid potato cultivar. Nat Genet 54:342–348.
夏瑞,华南农业大学教授,博士生导师;2013年获得美国弗吉尼亚理工大学获得园艺学博士, 2019年获得广东省特支计划科技创新领军人才项目支持;2023年获得第六届“卫志明青年创新奖”。在Nature Genetics, Molecular Plant等学术刊物发表SCI论文60多篇,累计引用>10,000次,入选2022“中国高被引学者”(Elsevier)。目前主要利用生物信息学、基因组学及分子生物学等手段,围绕无患子科植物花性别分化机制以及岭南水果花果发育调控机理等生物学问题开展研究。
陈程杰,华南农业大学讲师,以基因组和生物信息学为主要研究手段,致力于香蕉枯萎病抗性机理解析。持续分享课题开展期间产生的新策略/新方法,形成生物软件并对外分发(含TBtools和GSAman)。在Molecular Plant和iMeta等学术刊物发表论文30余篇, 入选2022“中国高被引学者”。

