众人拾柴火焰高-荔枝基因结构注释人工校正完成
时间:2023-02-10 阅读量:598
>>>从一开始就觉得这是一个值得写下来的事情,本来上个学期末就想写,但是苦于这事那事, 没能整理成稿,趁今天有点感觉,赶紧码下来。<<<
基因组序列和注释的重要性对每个从事分子生物学相关研究的课题组不言而喻。基因组的大量测序,对分子方面的研究有了巨大的推动作用,但是附带产生了一些问题。例如,每次做基因克隆都要求学生先用RNAseq数据确认一下用于设计引物的mRNA转录本序列是否正确。其实,除模式生物(也不是100%正确)以外的大部分生物基因组中基因结构的注释都存在不少的问题,也就是大部分的基因结构注释是不准确的。因此,在设计引物做基因功能研究前是需要手工进行序列的确认的。
自然,荔枝基因的注释也不完美,虽然是我们自己弄的基因组,但每个注释流程都有它的缺陷。在平时的分析过程中,时不时会发现一些基因的结构注释存在问题。而且随着课题的深入,学生们碰到的问题越来越多,感觉到了“长痛不如短痛,需要解决问题”的时候。
正好几年前,CJ就在改造IGV的过程中,开发了IGV-GSAman,可以根据RNAseq数据来进行注释信息校正(应该当时就觉得Appollo太难用)。
既然到了“决战”时刻,那就行动起来,决定发动全课题组所有人员(包括老师)进行荔枝基因结构注释人工校正。10月19日(现在算是去年了)进行动员,10月21日开了启动“大会”,有Pizza,有饮料。

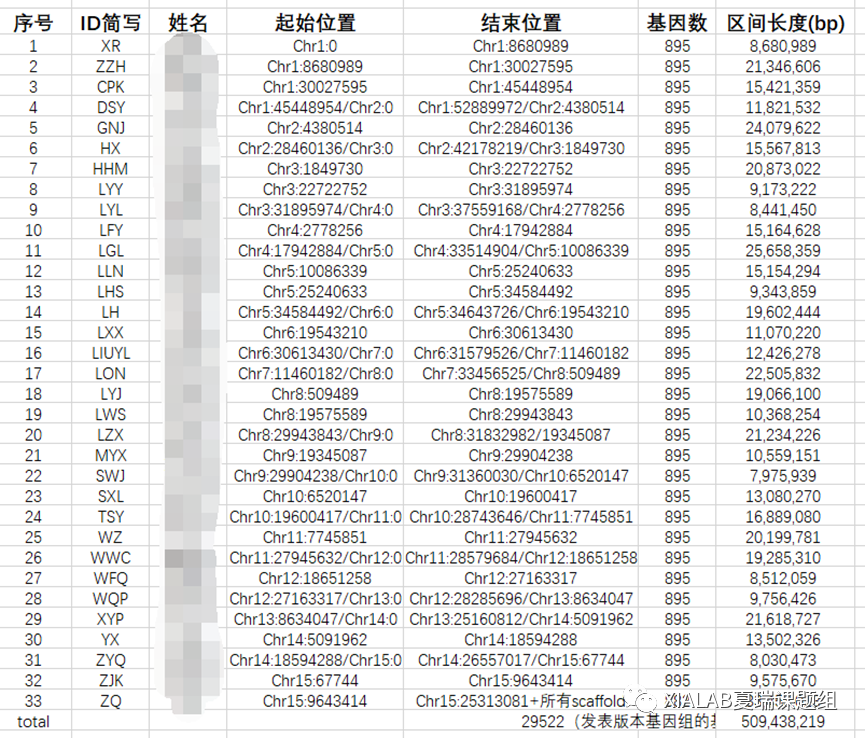
为体现公平公正,人人平等,每个人负责900来个基因,并计划一个月完成。
校正规则与要求要一致,比如UTR长度,转录起始与终止位置,内含子与外显子数目,转录本数量(重点关注最主要转录本m1),命名方式。。。

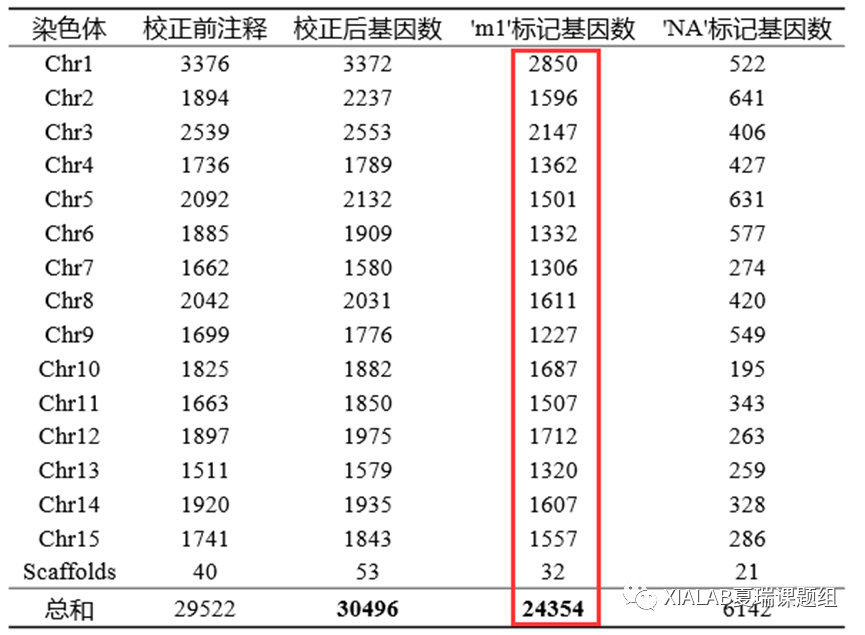
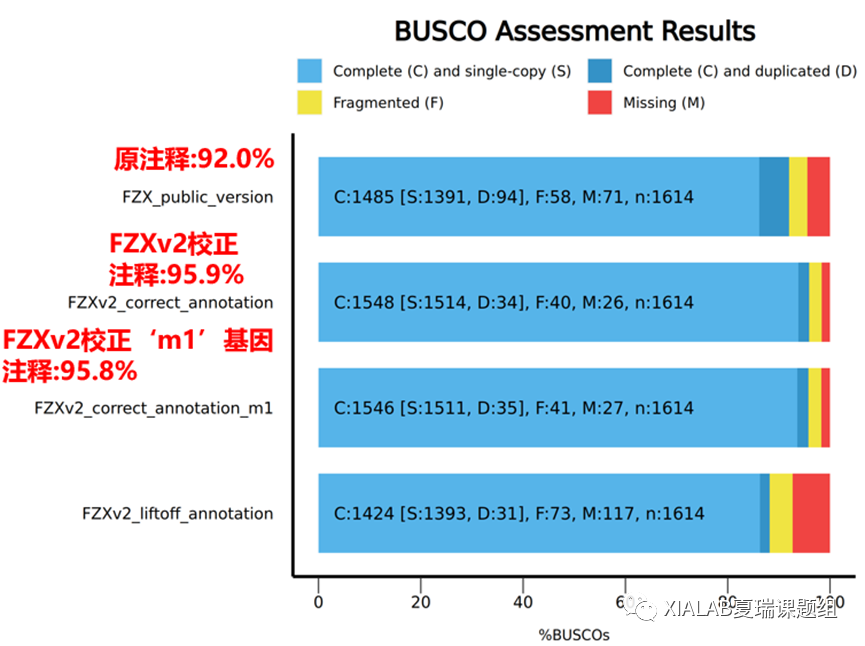
实际第一轮校正花了将近一个半月,12月初完成。校正后,荔枝基因结构注释质量明显提升,24,354个基因结构得到RNAseq数据证实和人工确认,注释完整度也从原本的92%提升到将近96%。
对于低年级学生,这是个学习的好机会,加深了对分子生物学书本知识的理解,也熟悉了IGV的使用;对于湿实验人员,学会了更好地利用测序和公共数据;对于干实验人员,好的注释有助于提升几乎所有的数据分析结果。
同时,在我们进行基因注释校正的过程中,不断根据实际需求,对GSAman工具提出修改意见,GSAman也更新了几十个版本(从19到72)。现在最新的版本较最初的版本,优化了不少功能,如快捷键,基于三代测序read直接生成转录本注释,注释校正实时保存功能。。。
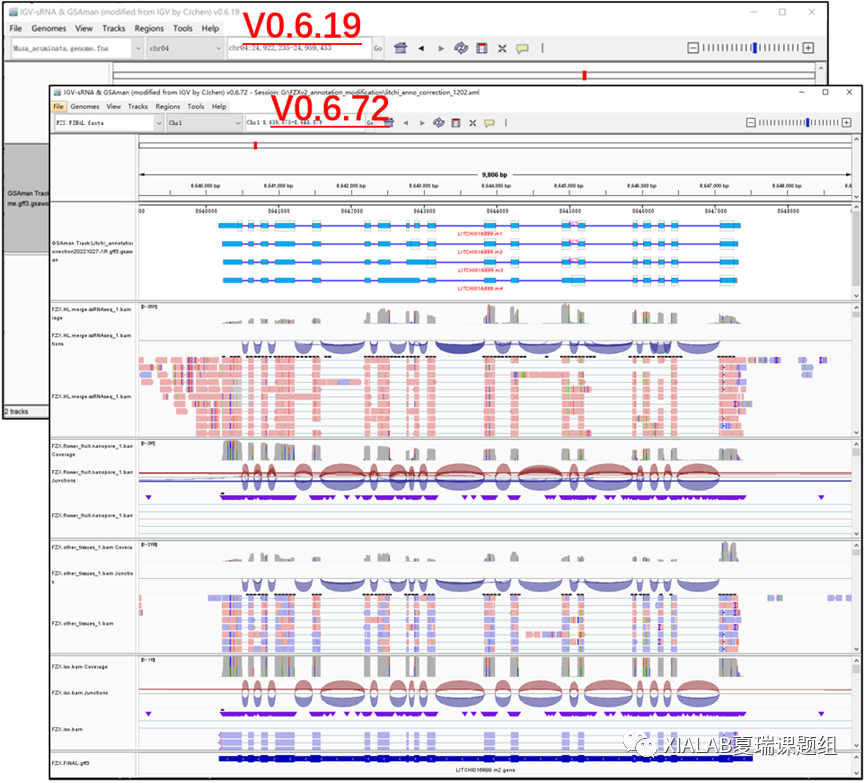
一个好的工具的开发,需要高超的技术开发能力的同时,也需要一线的对需求的细致理解。据说最新版的GSAman,一个人几天就可以校正完一个基因组了,似乎效能有了极大的提高。
总之,这是一项非常有意义的工作,也是对课题组后续研究工作有极大支撑作用的工作,虽然没有产生直接的研究成果,但是是一项基础性的一劳永逸的工作。
感谢课题组的每一个人!
课题组工作的成败靠所有人的共同努力!
PS:如果您也来自从事基因组或分子生物学相关研究的课题组,是PI,发动全课题组来一次全基因组注释校正(记得用IGV-GSAman),一劳永逸,还能练练兵;是学生,可以学知识,避免走弯路,也能为课题组留下永久性的贡献 :)
图文编辑:夏瑞

